Kapitel 10 Kontrollstrukturen
In diesem Kapitel sollen einige Prinzipien der Programmierung vorgestellt werden und gezeigt werden, wie sich diese in R umsetzen lassen. R bietet dabei eine Vielzahl an Möglichkeiten, von welchen einige besprochen werden sollen.
10.1 Prinzipien der Programmierung
R setzt in großen Teilen die Ideen der funktionalen Programmierung um. Das bedeutet für uns vor allem erst mal, dass ein Großteil der Problemlösung über Funktionen erfolgt. Folgende Prinzipien gilt es zu verinnerlichen, wenn man diese Eigenschaft effizient ausnutzen will:
DRY-Prinzip: “Don’t repeat yourself”. Treten wiederholt gleiche oder ähnliche Code-Elemente auf, so sollte man diese als Funktion zusammenfassen.
“Divide and Conquer”- Prinzip: Oft ist es besser, das Problem oder die Analyse in verschieden Teilprobleme aufzuteilen und für diese entsprechende Funktionen zu schreiben.
“Abstraktion”/Blackbox-Prinzip: Anstelle explizit viele Codezeilen im Hauptcode zu listen, welche letztlich gemeinsam eine bestimmte Funktion erfüllen, ist es besser, diese Komplexität in einer Funktion zu “verstecken”. Black Box: Entscheidend ist was die Funktion macht und nicht wie sie es macht.
Welche Möglichkeiten es gibt, diesen Prinzipien nachzukommen, wollen wir uns jetzt anschauen.
10.2 Funktionen selber schreiben
Funktionen waren schon oft unser Thema. Jetzt wollen wir uns anschauen, wie man selbst eine Funktion schreiben kann. Dies geschieht im Allgemeinen über dieses generelle Schema einer Funktionszuweisung:
- Mit der Funktion
function()können wir Funktionen erzeugen. :) Man beachte allerdings, dass nach den Klammern()noch etwas folgt. - Funktionen sind auch Objekte und können wie alle anderen Objekte auch an Namen gebunden werden.
- Eine Funktion hat typischerweise Argumentstellen (
arg1, ...), welche als sogenannte Funktionsparameter an denbodyweitergegeben werden. - Der
bodyoder auch Rumpf der Funktion enthält den Code, welcher beim Funktionsaufruf ausgeführt werden soll.
Eine konkrete Funktionszuweisung könnte dann so aussehen:
## [1] 4Die erzeugte Funktion nimmt eine Zahl x als Parameter entgegen und berechnet das Quadrat dieser. Da im body nichts Abweichendes definiert ist, wird das letzte Objekt zurückgegeben, also das Ergebnis unserer Berechnung. Unser Rückgabewert entspricht also dem Ergebnis der funktion x^2.
Optional kann man eine Rückgabe innerhalb des Rumpfes der Funktion auch erzwingen. Dazu verwendet man die Funktion return(). Zu beachten gilt es, dass die Ausführung der Funktion beim ersten Aufruf der return() Funktion abbricht und folgender Code nicht mehr berücksichtigt wird.
## [1] 4Im gegebenen Beispiel wird alles nach dem ersten return(y) nicht mehr ausgeführt. Der Rückgabewert der Funktion entspricht dem Wert des Objektes, welches mit dem Namen y verknüpft ist.
Wie wir gerade schon gesehen haben, können wir auch innerhalb von Funktionen Objekte an Namen binden. Dabei gilt es aber zu berücksichtigen, dass wir dabei kein Objekt in unserem Global Environment anlegen. Die Zuweisung gilt lediglich für den jeweiligen Wirkungsbereich (Scope) der Funktion, also alles, was in den geschweiften Klammern {...} steht.
## [1] 4## [1] 2Wie wir hier sehen können, bleibt die globale Zuweisung für den Namen x erhalten, trotz der erneuten Zuweisung eines Namens x innerhalb der Funktion.
Anmerkung:
Um den “Fluss” eurer Funktion nachzuverfolgen, könnt ihr euch Nachrichten auf der Konsole im Funktionsablauf ausgeben lassen. Das ist vor allem praktisch, wenn ihr euch nicht sicher seid, welchen Werte welches Objekt zu welchem Zeitpunkt hat.
x <- 2
quadrat <- function(x) {
message("Funktion beginnt mir ihrer Arbeit, x ist: ", x)
x <- x^2
message("Erste Berechnung erfolgreich, x ist: ", x)
x
}
quadrat(x)## [1] 4Alternativ könnt ihr eurer Variablen x einen konstanten Wert zuweisen, zum Beispiel x <- 2, und den {body} der Funktion Schritt für Schritt ausführen.
Wenn man einen sogenannten Default-Wert hinterlegt, dann kann eure Funktion auch ohne eine Belegung der entsprechenden Argumentstelle aufgerufen werden:
## [1] 4## [1] 100Defaults können sehr nützlich sein, um Komplexität zu verstecken.
Seit R 4.1.0 gibt es in R einen Shortcut um anonyme Funktionen zu deklarieren:
## [1] 8Statt function schreibt man einfach \ und die Klammern des Rumpfes {} werden einfach weggelassen. Das ist vor allem dann sinnvoll, wenn man eine Funktion ad hoc deklarieren will, zum Beispiel als Argument einer anderen Funktion. Darauf kommen wir später noch einmal zurück. Allerdings kann diese verkürzte Schreibweise auch zur schlechteren Lesbarkeit eures Codes führen.
10.3 Kontrollstrukturen
Kontrollstrukturen dienen der Ablaufsteuerung und sind essenziell für die Konstruktion von Algorithmen:
Folgende Logiken können dabei implementiert werden:
- Verzweigungen/Fallunterscheidungen:
- Falls heute die Sonne scheint, geh in den Park. Falls nicht, dann lerne für die nächste Klausur.
- Schleifen:
Solange die Sonne scheint, bleibe im Park.
Bestimme alle Quadratzahlen von \(1^2\) bis \(100^2\).
Ein klassische Fallunterscheidung können wir in R mit einer if-else Abfolge umsetzen:
## [1] "1 ist positiv"Wir setzen hier zunächst x als 1. Danach kommt die logische Abfrage, welche entweder TRUE oder FALSE sein muss. Ist das Ergebnis TRUE, dann wird der Code in den ersten geschweiften Klammern {print(paste(x, "ist positiv"))} ausgeführt und die Auswertung bricht ab. Falls das Ergebnis FALSE ist, wird der Code in den ersten geschweiften Klammern übersprungen und der else Fall wird ausgeführt.
Alternativ können wir if-else auch ohne geschweift Klammern schreiben:
## [1] "1 ist positiv"Hierbei gilt es allerdings zu beachten, dass, wenn wir nur die erste Zeile (if (x > 0)) ausführen, der restliche Code erst mal nicht berücksichtigt wird. Wir erhalten eine partiell ausgeführte Funktion zurück, welche noch weitere Eingaben erwartet. Führen wir die folgenden beiden Zeilen aus, kommen wir zum selben Ergebnis. Was zunächst umständlich erscheint, ist im body einer Funktion kein Problem, da dieser sowieso von oben nach unten durchläuft.
Ein Beispiel für eine Funktion mit Fallunterscheidung könnte so aussehen:
## [1] FALSE## [1] TRUEEs mal ohne geschweifte Klammern umzuschreiben wäre eine gute Übung!
10.3.1 Vektorisierte Fallunterscheidungen
Wollen wir if-else auf einen Vektor anwenden und erwarten einen Vektor als Ergebnis, müssen wir keine eigene Funktion schreiben. Es existieren bereits vektorisierte if-else Funktionen, zum Beispiel aus dem Package dplyr:::
library(dplyr)
x <- c(1991, 2004, 1988)
if_else(condition = x >= 1981 & x <= 1996,
true = "Millennial",
false = "kein Millennial")## [1] "Millennial" "kein Millennial" "Millennial"Wenn ihr mehr als zwei Fälle (TRUE und FALSE) unterscheiden wollt, bietet sich die ebenfalls vektorisierte dplyr::case_when() Funktion an. Eine Beispielanwendung dafür könnte die Kategorisierung einer stetigen Variable sein:
starwars %>%
select(name, birth_year) %>%
mutate(
age_categorized = case_when(
birth_year > 200 ~ "stone_old",
birth_year <= 200 & birth_year > 100 ~ "old",
birth_year <= 100 & birth_year > 20 ~ "mid_aged",
birth_year <= 20 ~ "young",
TRUE ~ "other" # catch them all
)
)## # A tibble: 87 × 3
## name birth_year age_categorized
## <chr> <dbl> <chr>
## 1 Luke Skywalker 19 young
## 2 C-3PO 112 old
## 3 R2-D2 33 mid_aged
## 4 Darth Vader 41.9 mid_aged
## 5 Leia Organa 19 young
## 6 Owen Lars 52 mid_aged
## 7 Beru Whitesun Lars 47 mid_aged
## 8 R5-D4 NA other
## 9 Biggs Darklighter 24 mid_aged
## 10 Obi-Wan Kenobi 57 mid_aged
## # ℹ 77 more rowsAnmerkung: Falls euch dieses Zeichen unbekannt vorkommt: %>% dann schaut doch noch mal in das Kapitel 5, dort haben wir Pipes mit der analogen base-R Notation (|>) eingeführt.
Im Beispiel wird eine neue Spalte age_categorized angelegt, welche eine Kategorisierung des Geburtsalters darstellt. Die einzelnen Fälle, welche in case_when() beschrieben werden, sind dabei jeweils durch ein Komma , getrennt. Vor der Tilde ~ kommt immer die Bedingung. Dahinter steht, was in diesem Fall passieren soll. Die letzte Bedingung TRUE ist immer wahr und greift dann, wenn alle vorherigen Bedingungen zu FALSE evaluiert worden sind. In unserem Beispiel fallen darunter alle NA-Werte. Die Rückgabe ist ein character-Vektor derselben Länge wie der von der Variable birth_year.
10.4 Schleifen
Schleifen (Loops) dienen dem wiederholten Ausführen eines Anweisungsblocks (loop body). Es gibt sie in verschiedenen Varianten:
for (i in I) {Anweisung}
- Interpretation: Wiederhole Ausdruck für jeden Indexwert
iin der IndexmengeI.
while (Bedingung) {Anweisung}
- Intepretation: Wiederhole
Anweisung, solange Bedingung erfüllt ist.
repeat {Anweisung}
- Interpretation: Wiederhole die Anweisung.
Anmerkung: Zusätzlich lassen sich diese Loops steuern mit next (springt zur nächsten
Iteration) und break (bricht die Schleife ab).
Wir werden uns hier nur noch der wohl gängigsten Methode, nämlich des for-loops, widmen. Schauen wir uns zunächst das einfachste Beispiel für einen for-loop an:
## [1] 1
## [1] 2
## [1] 3
## [1] 4
## [1] 5
## [1] 6
## [1] 7
## [1] 8
## [1] 9
## [1] 10Der for-loop iteriert über einen Vektor mit den Zahlen 1 bis 10 und führt den Code in den geschweiften Klammern in jeder Iteration aus. Der Index i wird nach jedem Schleifendurchlauf um eine Position inkrementiert.
Mit diesem Wissen können wir nun einen for-loop schreiben, welcher über die Werte eines Vektors iteriert und jeweils den Wert der i’ten Position im Quadrat in einen neuen Vektor schreibt.
v1 <- 1:10
v2 <- numeric(length = 10) # leeren Vektor anlegen
for (i in 1:10){
v2[i] <- sqrt(v1[i])
}
v2## [1] 1.000000 1.414214 1.732051 2.000000 2.236068 2.449490 2.645751 2.828427
## [9] 3.000000 3.162278Da die meisten Funktionen in R bereits vektorisiert sind (sqrt(v1)) ist ein for-loop nicht immer die beste Variante, manchmal kann er allerdings sehr hilfreich sein.
10.5 Vektorisiertes Programmieren
Vektorisiertes Programmieren bedeutet, dass wir Operationen auf einem Vektor ausführen und dementsprechenden einen Vektor zurückbekommen. Dabei wird eine Funktion auf jedes Element eines Vektors angewandt, was auch eine Möglichkeit ist, iterative Probleme zu lösen. Im letzten Teil wollen wir uns jetzt noch die purrr::map() Funktionsfamilie anschauen, welche vektorbasiert funktioniert. Alternativ dazu gibt es im base-R die lapply() Funktionsfamilie, welche eine ähnliche Intention verfolgt, die wir uns aber nicht mehr anschauen werden können.
Die Aufgabe, welche wir gerade mit einem for-loop gelöst haben, kann man alternativ auch vektorbasiert mit map() lösen:
## [1] 1.000000 1.414214 1.732051 2.000000 2.236068 2.449490 2.645751 2.828427
## [9] 3.000000 3.162278Dabei passiert Folgendes (fun steht für function):
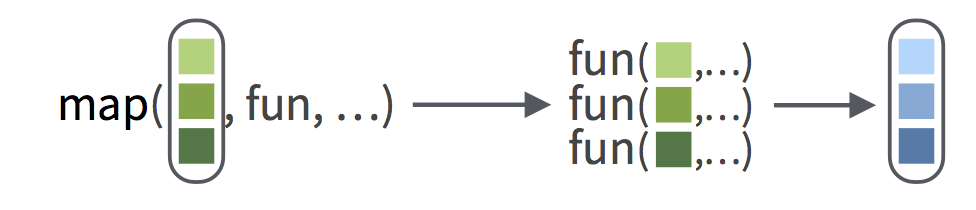
Abb. 10.1: Quelle: https://tutorials.shinyapps.io/map-intro/_w_543ccdaf/www/images/map.png
Generell sind die map() Funktionen nach diesem Schema aufgebaut:
Wichtig!: Wenn wir eine Funktion als Objekt übergeben, setzen wir keine Klammern.
Der Rückgabetyp, welchen wir oben im Beispiel verwendet haben, war dbl, was einer Fließkommazahl, also im Prinzip numeric entspricht. Als erstes Argument übergibt man eine Liste oder besser gesagt einen Vektor. Die Liste ist eigentlich nur ein Sonderfall von verschachtelten Vektoren, weshalb wir auch einen Vektor übergeben können. Richtig nützlich wird map() aber erst, wenn man es mit Listen zu tun hat. Das zweite Argument ist eine Funktion, welche über ihren Namen als an map() übergeben wird.
Benutzen wir die map() Funktion ohne Rückgabetyp, bekommen wir immer eine Liste zurück:
## [[1]]
## [1] 0
##
## [[2]]
## [1] 0.6931472
##
## [[3]]
## [1] 1.098612Was für Funktionen es alles gibt, ist unter anderem auf dem Cheatsheet zu purrr:: sehr übersichtlich dargestellt.
Es ist auch möglich, der map() übergebenen Funktion noch weitere Argumente zu übergeben. Diese werden map() einfach als weitere Argumente übergeben:
## [1] 0.00 0.69 1.10 1.39 1.61 1.79 1.95 2.08 2.20 2.30Über sogenannte anonyme Funktionen (welche keinen Namen haben), können wir gleich in der Argumentstelle von map(), welche die Funktion entgegennimmt, eigene Funktionen schreiben.
Beispiel:
## [1] 0.00 0.69 1.10 1.39 1.61 1.79 1.95 2.08 2.20 2.30Die Tilde (~) zeigt dabei an, dass eine Funktion folgt. Mit .x kann man das aktuelle Listenelement ansprechen. Ausgeschrieben könnte die Funktion so ausschauen:
Natürlich könnt ihr die Funktionen auch erst schreiben und dann nur noch über den Namen map() als Argument weiter reichen.
Dem Beispiel folgend:
## [1] 0.00 0.69 1.10 1.39 1.61 1.79 1.95 2.08 2.20 2.30Bei diesem Vorgehen empfiehlt es sich, einen aussagekräftigeren Namen für die Funktion zu wählen, welcher auch andeutet, was die Funktion macht.
10.7 Literaturverweise
Ergänzend
- R for Data Science Kapitel 19 (Functions)
- Hands-On Programming with R Kapitel 2.4 bis 2.7
Weiterführend
- Advanced R Kapitel 9 (Functional Programming)